Activités de recherches
Les enzymes sont des biomolécules capables de catalyser un grand nombre de réactions chimiques, en limitant l’utilisation de solvants organiques et en opérant dans des conditions douces (température, temps de réaction…). Dans l’équipe Enzymologie et Glycobiochimie, deux familles d’enzymes sont principalement utilisées, qui sont naturellement impliquées dans la synthèse (Glycosyltransférases) ou la dégradation (Glycoside Hydrolases) des sucres.
Notre approche méthodologique, liant le fondamental à l’applicatif, est décomposée en 3 parties :

1. Identification et caractérisation de nouvelles enzymes d’intérêt en biocatalyse.
En s’appuyant sur des approches combinant la biologie moléculaire (analyse génomique, clonage, …), la biochimie des protéines (expression, purification), l’enzymologie (biocatalyse, cinétique, …) et la biologie structurale (Modélisation moléculaire, cristallographie …), des enzymes candidates sont initialement identifiées, produites, et caractérisées. Leur sélection est faite en s’appuyant sur les données d’annotation génomique, des caractéristiques recherchées, mais également de leur sélectivité de substrat potentielle (en choisissant par exemple des sucres rares).
3 exemples récents :
| α-L-Rhamnosidase (DtRha) Guillotin et al, 2019, ACS Omega | |
|
DtRha a été caractérisée comme pouvant hydrolyser sélectivement les groupements rhamnosyles de flavonoïdes naturels, ouvrant la voie à la synthèse de métabolites intermédiaires. |
|
| β-D-Galactofuranosidase (Galf-ase) – Seničar et al, 2019, Carb Res. | |
|
Galf-ase est la première galactofuranosidase clonée spécifique de ce sucre rare (galactofuranose), retrouvé à la surface cellulaire de nombreux pathogènes. |
 |
| Galactofuranosyltransférases LPG1 – Ati et al, 2018, Sci Rep. | |
|
Les 4 galactofuranosyltransférases LPG1 ont été produites à partir du génome du pathogène Leishmania major, et ont démontré une spécificité de substrat originale et ouvrant des perspectives intéressantes en synthèse. |
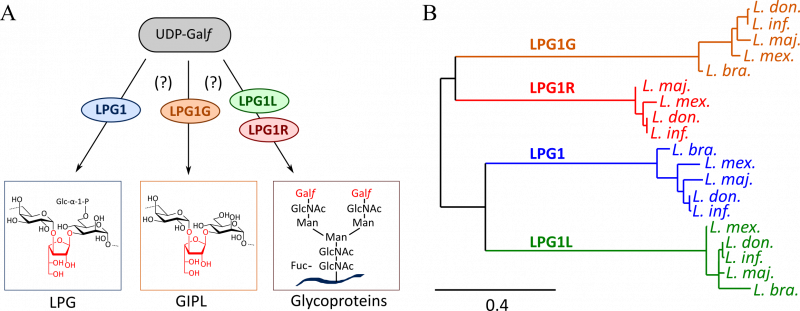 |
2. Ingéniérie des Protéines
L’ingénierie génétique (mutagenèse dirigée, ...) permet de moduler à façon la séquence peptidique des enzymes. Dans l’équipe Enzymologie et Glycobiochimie, nous utilisons cette approche avec 2 objectifs : pouvoir comprendre finement le mécanisme d’action de ces enzymes, mais également de modifier l’activité de ces enzymes pour obtenir des biocatalyseurs plus efficaces.
3 exemples récents :
| S-Glycosyltransférase UGT74B1 – Lafite et al., 2019, Mol Cat | |
|
L’activité de UGT74B1 est correllée à l’acidité du thiol qui réagit. La mutagenèse dirigée de plusieurs résidus du site actif a permis d’identifier le mécanisme de cette réaction originale. |
|
| β-D-Glucuronidase DtGlcA – Kurdziel et al, 2020, Org. Biomol. Chem. | |
|
L’inactivation par mutagenèse d’un l’acide aminé catalytique des glycoside hydrolase permet d’obtenir une nouvelle activité de synthèse de S-glycosides : les thioligases. Cette méthodologie a été appliquée à plusieurs enzymes au laboratoire, dont la b-D-glucuronidase DtGlcA. |
 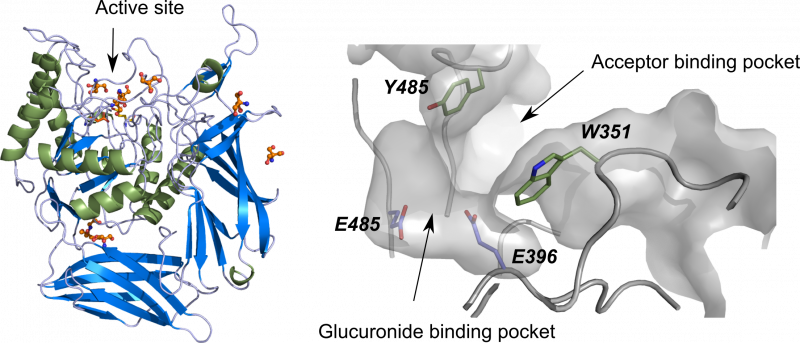 |
| β-D-Glycosidase DtGly– Guillotin et al, 2019, Catalysts | |
|
L’utilisation de donneurs de sucres originaux (Thioimidates) par DtGly a été étudiée, et l’étude de mutants a permis de comprendre le mécanisme de cette réaction. |
 |
3. Un exemple d’application : la préparation de O-glycosides comme conservateurs en cosmétique (Guillotin et al., 2017, Pure and Applied Chem.).
L’utilisation comme biocatalyseur d’une enzyme clonée à partir de Dictyoglomus thermophilum a permis la préparation d’une série de dérivés glycosylés du glycérol, qui ont montré une activité antimicrobienne vis-à-vis de souches d’Aspergillus en micro-challenge test.











